-
Mental相关性网路图 | 环形网路图 | 二分式网路图
相关性网络图
date: 2023.10.12
教程收集与整理: 小杜的生信笔记
一边学习,一边总结,一边分享!

本期教程图形

写在前面
相关性分析,在前期教程中也有提及。但是使用R语言绘制相关性网络图,除了使用
ggcor包做Meantal分析外,就没有类似的教程。本期教程,自己也收集相关的包和教程,分别绘制对应的图形。本期教程,也是值得大家收藏,现在的代码基本是直接粘贴复制即可。
1 安装和加载相关的R包
library(ggraph) library(tidygraph) # install.packages("devtools") #devtools::install_github("Hy4m/linkET", force = TRUE) library("linkET") packageVersion("linkET") packageVersion("igraph") #devtools::install_github("Hy4m/netET") library(netET)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
设置路径
setwd("E:\\小杜的生信筆記\\2023\\20231012-mental分析网络图")- 1
2 加载数据
matrix_data(list(mtcars = mtcars)) matrix_data(list(mtcars = mtcars)) %>% as_md_tbl() as_matrix_data(mtcars) head(mtcars)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
3 计算r值和p值
as_md_tbl(mtcars) correlate(mtcars) %>% as_md_tbl()- 1
- 2
- 3
- 4
4 绘图
4.1 绘制组内相关性热图
组内相关性分析,我们在前面的教程重要发表过,详情可以看R语言可视化-精美图形绘制系列–组内相关性分析。我们也提供完整的输出P值和cor值的代码。
correlate(mtcars) %>% as_md_tbl() %>% qcorrplot() + geom_square()- 1
- 2
- 3
- 4
前期教程代码,计算组内相关性
library(reshape2) library(corrplot) library(plyr) library(igraph) library(autoReg) library(tidyverse) library(ggsci) library(stats) ## 详情可以到此教程中查看 corr <- cor(mtcars, method = "spearman")- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
corrplot(corr,title = "", method = "circle", #或"circle" (default), "square", "ellipse", "number", "pie", "shade" and "color" outline = T, addgrid.col = "darkgray", order="hclust", addrect = 4, #hclust聚为4类,根据数据的具体情况调整 mar = c(4,0,4,0), rect.col = "black", rect.lwd = 2, cl.pos = "b", tl.col = "black", tl.cex = 1, cl.cex = 1.5, tl.srt=60)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
corrplot(corr,order = "AOE",type="upper",tl.pos = "tp") corrplot(corr, title = "", method = "number", outline = T, add = TRUE, type = "lower", order="AOE", # col="black", # diag=FALSE, tl.pos="n", cl.pos="n") ## 注意:此步骤在R MarkDown格式中运行报错,但在非R MarkDown格式中可以正常运行- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
输出相关性p值和corr值代码如下:

4.1 绘制组间相关热图
同样组内相关分析分析,在前期教程中也发布过。详情请看R语言可视化-精美图形绘制系列–组间相关性分析。
4.2.1 加载数据
首先,使用教程中的代码进行计算,运行。
library(vegan) data("varespec") data("varechem")- 1
- 2
- 3
dim(varespec) varespec[1:10,1:10]- 1
- 2
- 3
查看数据
dim(varechem) varechem[1:10,1:10]- 1
- 2
4.2.2 计算两数据的相关性
绘制相关性热图
correlate(varespec[1:30], varechem) %>% qcorrplot() + geom_square() + scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu"))- 1
- 2
- 3
- 4

qcorrplot(varespec[1:30], type = "lower") + geom_square() + scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu"))- 1
- 2
- 3

5 进行mantel分析
mantel相关性分析,早期教程ggcor包作图 | 相关性热图 | mental分析图。
那么使用
linkET包,也是非常的方便。5.1 加载数据
##mantel test library(dplyr) data("varechem", package = "vegan") data("varespec", package = "vegan")- 1
- 2
- 3
- 4
- 5
5.2 查看数据
## 查看数据 dim(varespec) # [1] 24 44 varespec[1:10,1:10]- 1
- 2
- 3
- 4
dim(varechem) # [1] 24 14 varechem[1:10,1:10]- 1
- 2
- 3
5.3 计算网络关系
mantel <- mantel_test(varespec, ## 分类数据 varechem, ## 影响因子数据 ## 以下代码是根据varespec(分类数据)进行分析计算 spec_select = list(Spec01 = 1:7, Spec02 = 8:18, Spec03 = 19:37, Spec04 = 38:44)) %>% mutate(rd = cut(r, breaks = c(-Inf, 0.2, 0.4, Inf), labels = c("< 0.2", "0.2 - 0.4", ">= 0.4")), pd = cut(p, breaks = c(-Inf, 0.01, 0.05, Inf), labels = c("< 0.01", "0.01 - 0.05", ">= 0.05")))- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
查看数据
head(mantel) ### > head(mantel) # A tibble: 6 × 6 spec env r p rd pd1 Spec01 N 0.256 0.015 0.2 - 0.4 0.01 - 0.05 2 Spec01 P 0.137 0.093 < 0.2 >= 0.05 3 Spec01 K 0.400 0.004 >= 0.4 < 0.01 4 Spec01 Ca 0.0113 0.427 < 0.2 >= 0.05 5 Spec01 Mg 0.0263 0.366 < 0.2 >= 0.05 6 Spec01 S 0.275 0.021 0.2 - 0.4 0.01 - 0.05 - 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
5.3 绘制mantel分析图
## 绘制相关性热图 D0 <- qcorrplot(correlate(varechem), type = "lower", diag = FALSE) + geom_square() + ## 相关性热图的形状 ## geom_couple(aes(colour = pd, size = rd), data = mantel, curvature = nice_curvature()) + ## 颜色参数调整 scale_fill_gradientn(colours = RColorBrewer::brewer.pal(11, "RdBu")) + scale_size_manual(values = c(0.5, 1, 2)) + scale_colour_manual(values = color_pal(3)) + guides(size = guide_legend(title = "Mantel's r", override.aes = list(colour = "grey35"), order = 2), colour = guide_legend(title = "Mantel's p", override.aes = list(size = 3), order = 1), fill = guide_colorbar(title = "Pearson's r", order = 3)) D0 ggsave("Mental相关性网络图.jpg",width = 6, height = 6)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
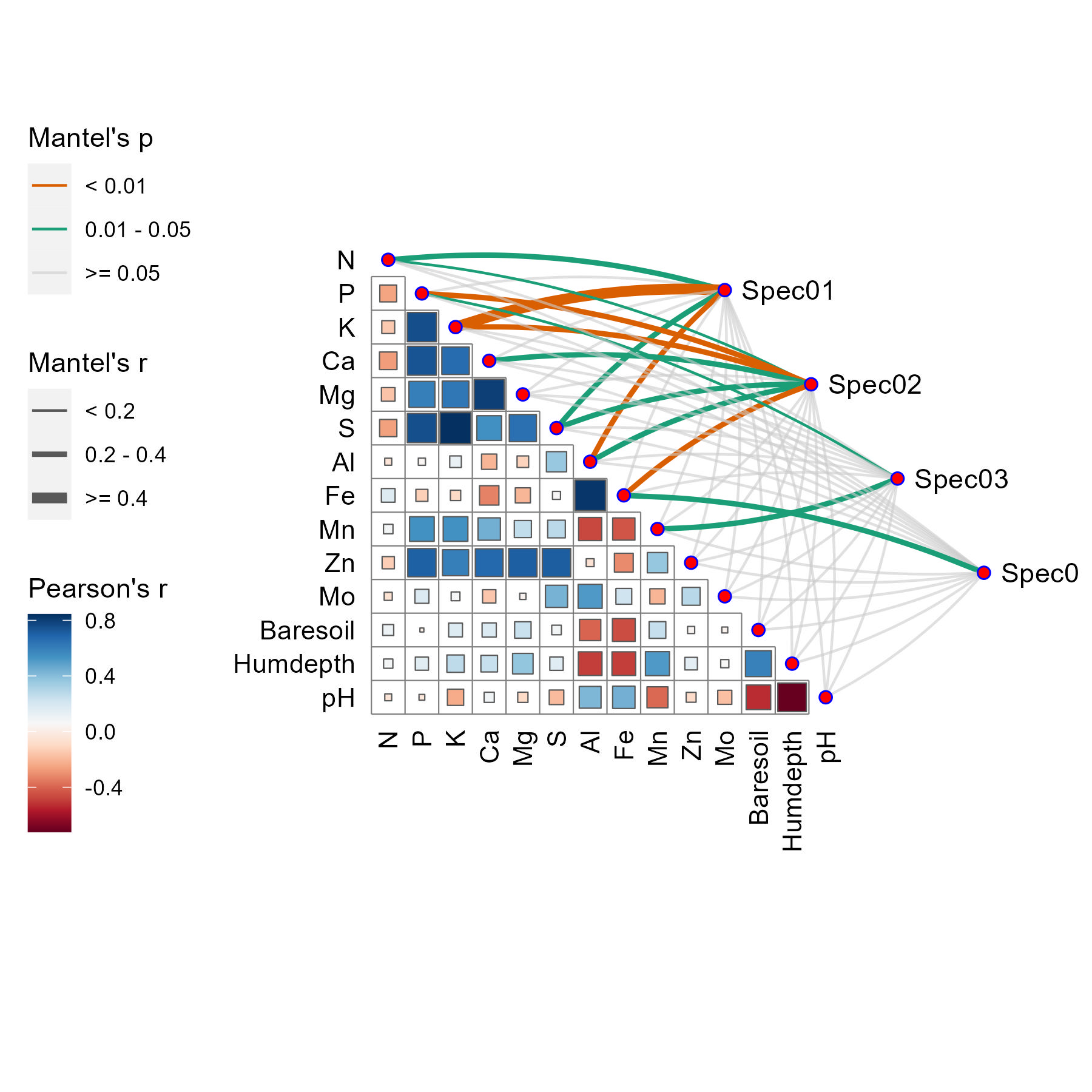
6 网络相关性图

此教程来自Lingc TONG的推文,原文教程链接基于netET和ggraph展示微生物与环境因子网络相关性。6.1 数据准备
这里依旧使用前面的数据即可。
devtools::install_github("Hy4m/netET", force = TRUE) #加载包 library(ggraph) library(tidygraph) library(netET)- 1
- 2
- 3
- 4
- 5
library(vegan) data("varespec") data("varechem")- 1
- 2
- 3
6.2 计算相关性,并提取绘图信息
p1 <- correlate(varechem, varespec, method = "spearman") |> as_tbl_graph(abs(r) > 0.5, p < 0.05) # 计算节点的度中心性 degree_centrality <- degree(p1) # 计算节点的度中心性 degree_centrality <- degree(p1, mode = "all") # 将中心性值添加到 p1 中 p1$Degree <- degree_centrality p1 <- p1 |> mutate(Degree = degree_centrality)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
注意:刚开始,使用原文的代码提取信息,一直报错。后面,提示
igraph包的版本过高,需要使用> 0.2版本包。在这里折腾了一会。最后依旧是降此包的版本。
步骤:- 删除已有的
igraph包 - 重新安装
igraph包,并限制版本
install.packages("igraph",version = '0.1.7')- 1
使用原文代码:
### 简洁代码 p1 <- correlate(varechem, varespec, method = "spearman") |> as_tbl_graph(abs(r) > 0.5, p < 0.05) |> mutate(Degree = centrality_degree())- 1
- 2
- 3
- 4
> p1 # A tbl_graph: 58 nodes and 30 edges # # An undirected simple graph with 36 components # # A tibble: 58 × 2 name Degree1 N 2 2 P 1 3 K 1 4 Ca 2 5 Mg 2 6 S 0 # ℹ 52 more rows # ℹ Use `print(n = ...)` to see more rows # # A tibble: 30 × 4 from to r p 1 13 18 0.608 0.00163 2 1 20 -0.606 0.00168 3 7 26 -0.500 0.0128 # ℹ 27 more rows # ℹ Use `print(n = ...)` to see more rows - 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
xy <- layout_on_circle(p1) head(xy)- 1
- 2
> head(xy) [,1] [,2] [1,] 1.0000000 0.0000000 [2,] 0.9941380 0.1081190 [3,] 0.9766206 0.2149704 [4,] 0.9476532 0.3193015 [5,] 0.9075754 0.4198891 [6,] 0.8568572 0.5155539- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
6.3 绘制环形相关性图
D1 <- ggraph(p1, xy) + #geom_edge_fan(aes(colour = r > 0), width = 0.75, linetype="dashed") + #width 改变线条粗细 geom_edge_fan(aes(colour = r > 0), width = 0.8) + geom_node_point(aes(size = Degree), colour = "#fa8c35") + scale_edge_colour_manual(values = c("TRUE" = "#c93756", "FALSE" = "#21a675"),#R>0,为TRUE labels = c("Negative", "Positive")) + geom_node_text(aes(x = 1.07 * x, y = 1.07 * y, label = name, angle = node_angle(x, y)), hjust = "outward", data = function(data) dplyr::filter(data, Degree > 0)) + expand_limits(x = c(-1.5, 1.5), y = c(-1.5, 1.5)) + # coord_fixed(clip = "off") + theme(panel.background = element_blank()) + labs(edge_colour = "Spearman's r") ggsave("环形网络图.jpg", width = 6, height = 6)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18

6.4 二分网络图
p2 <- correlate(varechem, varespec, method = "spearman") |> as_tbl_graph(abs(r) > 0.5, p < 0.05) |> mutate(Degree = centrality_degree()) |> as_bipartite_circular(outer_nodes = names(varespec)) p2- 1
- 2
- 3
- 4
- 5
绘图
D2 <- ggraph(p2, layout_bipartite_circular(p2)) + annotate_arc_rect(0, 180, fill = "#e0eee8", r0 = 0.55, r1 = 1.05) + geom_edge_circular(aes(colour = r > 0), edge_width = 0.75, edge_alpha = 0.8) + geom_node_point(aes(size = Degree, colour = Degree == 0)) + geom_node_text_circular(expand = 0.08) + scale_colour_manual(values = c("TRUE" = "grey55","FALSE" = "#065279"), guide = "none") + scale_edge_colour_manual(values = c("TRUE" = "#c93756", "FALSE" = "#21a675"), labels = c("Negative", "Positive")) + coord_fixed(clip = "off", xlim = c(-1.2, 1.2), ylim = c(0, 1.1)) + theme(panel.background = element_blank()) + guides(edge_colour = guide_legend(override.aes = list(edge_width = 1))) + labs(edge_colour = "Spearman's r") D2 ggsave("二分网络图.jpg", width = 8, height = 8)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18

7 合并图形
library(patchwork) library(cowplot) D0+D1+D2+plot_layout(nrow = 1, ncol = 3, widths = c(6,5,6)) ggsave("20231012.jpg", width = 20, height = 10)- 1
- 2
- 3
- 4
- 5

往期文章:
1. 复现SCI文章系列专栏
2. 《生信知识库订阅须知》,同步更新,易于搜索与管理。
3. 最全WGCNA教程(替换数据即可出全部结果与图形)
4. 精美图形绘制教程
5. 转录组分析教程
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!
-
相关阅读:
javascript代码重构: 如何写好函数的9个技巧
Python 爬虫入门(十一):Scrapy高级应用之并发与分布式「详细介绍」
带你搞懂MySQL隔离级别,两个事务同时操作同一行数据会怎样?
iwebsec靶场 XXE关卡通关笔记
C++大神之路——环境篇
Leetcode 429:N叉树的层次遍历
浅浅探索Memcached
关于 Coremail windows邮箱客户端(Air 版)特定附件名称显示不正确问题的说明
INDICATOR 再c嵌入sql环境中的作用
js的数组如何根据元素内容删除
- 原文地址:https://blog.csdn.net/kanghua_du/article/details/133787128
