-
SOM网络2: 代码的实现
SOM自组织映射神经网络的原理,详见博客:SOM网络1:原理讲解
训练的主函数
train_SO代码如下:def train_SOM(X, # 输出节点行数 Y, # 输出节点列数 N_epoch, # epoch datas, # 训练数据(N x D) N个D维样本 init_lr=0.5, # 初始化学习率 lr sigma = 0.5, # 初始化 sigma 用来更新领域节点权重 dis_func = euclidean_distance, # 距离公式 默认欧拉距离 neighborhood_func = gaussion_neighborhood, # 邻域节点权重公式g 默认高斯函数 init_weight_fun=None, #初始化权重函数 seed=10): # 获取输入的特征维度 N,D =np.shape(datas) # 训练的步数 N_steps =N_epoch*N #对权重进行初始化 rng = np.random.RandomState(seed) if init_weight_fun is None: weights =rng.rand(X,Y,D)*2-1 #随机初始化 weights /=np.linalg.norm(weights,axis=-1,keepdims=True) #标准化 else: weights = init_weight_fun(X,Y,datas) # 一般使用PCA初始化- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
PCA 初始化权重
def weights_PCA(X,Y,data): N,D=np.shape(data) weights=np.zeros([X,Y,D]) pc_value,pc=np.linalg.eig(np.conv(np.transpose(data))) # pc_vale为特征值,pc 为特征向量 DXD维 pc_order=np.argsort(-pc_value) # 特征值从大到小排序,并返回Index # 对W:[X,Y,D]进行初始化 for i,c1 in enumerate(np.linspace(-1,1,X)): for j,c2 in enumerate(np.linsapce(-1,1,Y)): weights[i,j]=c1*pc[pc_order[0]]+c2*pc[pc_order[1]] #利用最大的2个特征值对应的特征向量加权组合成i,j位置的D维表征向量- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
完整的训练代码
def train_SOM(X, # 输出节点行数 Y, # 输出节点列数 N_epoch, # epoch datas, # 训练数据(N x D) N个D维样本 init_lr=0.5, # 初始化学习率 lr sigma = 0.5, # 初始化 sigma 用来更新领域节点权重 dis_func = euclidean_distance, # 距离公式 默认欧拉距离 neighborhood_func = gaussion_neighborhood, # 邻域节点权重公式g 默认高斯函数 init_weight_func=weights_PCA, #初始化权重函数 seed=10): # 获取输入的特征维度 N,D =np.shape(datas) # 训练的步数 N_steps =N_epoch*N #对权重进行初始化 rng = np.random.RandomState(seed) if init_weight_func is None: weights =rng.rand(X,Y,D)*2-1 #随机初始化 weights /=np.linalg.norm(weights,axis=-1,keepdims=True) #标准化 else: weights = init_weight_fun(X,Y,datas) # 一般使用PCA初始化 for n_epoch in range(N_epoch): print("Epoch %d" %(n_epoch+1)) #打乱样本次序 index=rng.permulation(np.arange(N)) for n_step,_id in enumerate(index): # 取一个样本 x=datas[_id] #计算learning rate (eta) t=N*n_epoch + n_step eta=get_learning_rate(init_lr,t,N_steps) #计算样本距离输出的每个节点的距离,并获取激活点的位置 winner=get_winner_index(x,weights,dis_func) #根据激活点的位置计算临近点的权重 随着迭代的进行sigma也需要不断减少 new_sigma=get_learning_rate(sigma,t,N_steps) # sigma 更新的方式和学习率一样 g=neighborhood_fun(X,Y,winner,new_sigma) g=g*eta #进行权重的更新 weights = weights + np.expand_dims(g,-1)*(x-weights) # 打印量化误差 print("quantization_error=%.4f" %(get_quantization_error(data,weights))) return weights #计算学习率 def get_learning_rate(lr,t,max_steps): # t当前的steps max_steps=N x epoch (N样本数) return lr/(1+t/(max_steps/2)) # 获取激活(获胜点)节点的位置,与x距离最小的输出节点位置 def get_winner_index(x,w,dis_func=euclidean_distance): # 计算输入样本和各个节点的距离 dis = dis_func(x,w) #找到距离最小的位置 index=np.where(dis ==np.min(dis)) return (index[0][0],index[1][0]) #利用高斯距离法计算临近点的权重 # X,Y模板大小,c中心点的位置 def gaussion_neighborhood(X,Y,c,sigma) xx,yy=np.meshgrid(np.arange(X),np.arange(Y)) d=2*sigma*sigma ax=np.exp(-np.power(xx-xx.T[c],2)/d) ay=np.exp(-np.power(yy-yy.T[c],2)/d) return (ax*ay).T # 计算欧式距离 def euclidean_distance(x,w): dis=np.expand_dims(x,axis=(0,1))-w # x:D w:[X,Y,D] 因此需要增加两维 x:D->x:[1,1,D] return np.linalg.norm(dis,axis=-1) # 输出[X,Y] 二范数 即为欧拉距离 # 特征标准化 (x-mu)/std def feature_normalization(data): mu=np.mean(data,axis=0,keepdims=True) sigma=np.std(data,axis=0,keepdims=True) return (data-mu)/sigma def get_U_Matrix(weights): X,Y,D=np.shape(weights) um=na.nan * np.zeros((X,Y,8)) #8 领域 ii=[0 ,-1,-1,-1,0,1,1, 1] jj=[-1,-1, 0, 1,1,1,0,-1] for x in range(X): for y in range(Y): w_2=weights[x,y] for k,(i,j) in enumerate(zip(ii,jj)): if(x+i >=0 and x+i<X and y+j>=0 and y+j <Y): w_1=weights[x+i,y+j] um[x,y,k]=np.linalg.norm(w_1-w_2) um=np.nansum(um,axis=2) return um/um.max() #计算量化误差 计算每个样本点和映射点之间的平均距离 def get_quantization_error(data,weights): w_x,w_y=zip(*[get_winner_index(d,weights) for d in datas]) error=datas-weights[w_x,w_y] # 数据域聚类中心的距离 error=np.linalg.norm(error,axis=-1) return np.mean(error)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 56
- 57
- 58
- 59
- 60
- 61
- 62
- 63
- 64
- 65
- 66
- 67
- 68
- 69
- 70
- 71
- 72
- 73
- 74
- 75
- 76
- 77
- 78
- 79
- 80
- 81
- 82
- 83
- 84
- 85
- 86
- 87
- 88
- 89
- 90
- 91
- 92
- 93
- 94
- 95
- 96
- 97
- 98
- 99
- 100
- 101
- 102
- 103
- 104
- 105
- 106
- 107
训练完成后,返回输出节点的
weights,维度为 [ X , Y , D ] [X,Y,D] [X,Y,D], 相当于固化了模型的权重weights,weights表征了当前的训练样本。测试
if __name__ == "__main__": # seed 数据展示 columns=['area','perimeter','compactness','length_kernel','width_kernel', 'asymmetry_coefficient','length_kernel_groove','target'] data = pd.read_csv('seeds_dataset.txt',names=columns,sep='\t+',engine='python') labs=data['target'].values lab_names={1:'Kama',2:'Rosa',3:'Canadian'} datas=data[data.columns[:-1]].values N,D=np.shape(datas) print(N,D) # 对训练数据进行标准化 datas = feature_normalization(datas) #SOM的训练 weights=train_SOM()X=9,Y=9,N_epoch=2,datas=datas,sigma=1.5,init_weight_func=weights_PCA) # 获取UMAP 用于可视化 UM=get_U_Matrix(weights) plt.figure(figure=(9,9)) plt.pcolor(UM.T,cmap='bone_r') #plotting the distance map as background plt.colorbar()- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
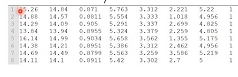
测试数据
U_Matrix
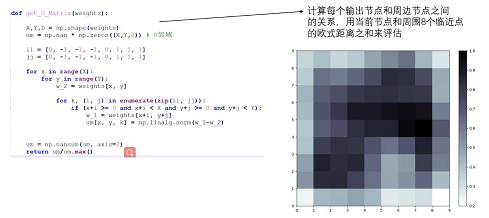
- 颜色越深说明与邻近点的关系越强烈,颜色越强说明与邻近点的关系越不强烈。
测试分类的效果
```python if __name__ == "__main__": # seed 数据展示 columns=['area','perimeter','compactness','length_kernel','width_kernel', 'asymmetry_coefficient','length_kernel_groove','target'] data = pd.read_csv('seeds_dataset.txt',names=columns,sep='\t+',engine='python') labs=data['target'].values lab_names={1:'Kama',2:'Rosa',3:'Canadian'} datas=data[data.columns[:-1]].values N,D=np.shape(datas) print(N,D) # 对训练数据进行标准化 datas = feature_normalization(datas) #SOM的训练 weights=train_SOM()X=9,Y=9,N_epoch=2,datas=datas,sigma=1.5,init_weight_func=weights_PCA) # 获取UMAP 用于可视化 UM=get_U_Matrix(weights) plt.figure(figure=(9,9)) plt.pcolor(UM.T,cmap='bone_r') #plotting the distance map as background plt.colorbar() # 查看分类的效果 markers=['o','s','D'] colors =['C0','C1','C2'] for i in range(N): x =datas[i] w=get_winner_index(x,weights) i_lab=labs[i]-1 plt.plot(w[0]+.5,w[1]+.5,markers[i_lab],markerfacecolor='None' markeredgecolor=colors[i_lab],markersize=12,markeredgewidth=2) plt.show()- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36

-
相关阅读:
TensorRT基础笔记
Redis知识点复习
【算法心得】minus instead of add
【图像处理】关于颜色的万花筒(RGB--HSV)
HTML+CSS+JS宠物商城网页设计期末课程大作业 web前端开发技术 web课程设计 网页规划与设计
AP8851H 宽电压降压恒压DC-DC 电源管理芯片 5V 12V输出 零功耗 快充方案应用
Redis 5 种基本数据结构(String、List、Hash、Set、Sorted Set)详解 | JavaGuide
数据通讯基础
canvas绘制刮涂层抽奖效果
06 Spring_AOP
- 原文地址:https://blog.csdn.net/weixin_38346042/article/details/126100743
